Michael Palmer, MD y Jonathan Gilthorpe, PhD
Estudios recientes realizados por Kevin McKernan, un destacado experto en métodos de secuenciación de ADN y ARN, han revelado que los lotes de vacunas de ARNm modificadas producidas tanto por Pfizer como por Moderna contienen una alta proporción de ADN bacteriano contaminante. En total, el ADN representa hasta un 20-35% de los ácidos nucleicos contenidos en cada uno de los lotes de vacunas. Estas concentraciones alarmantemente altas superan con creces los niveles considerados seguros por organizaciones normativas como la Agencia Europea de Medicamentos (EMA). Este documento resume la evidencia de esa contaminación de ADN y discute los posibles riesgos para la salud que implica para los receptores de las vacunas.
1. El papel del ADN en la fabricación de vacunas de ARNm
1.1. Antecedentes generales
La mayoría de los lectores sabrán que
- los ARN sintéticos contenidos en las vacunas de ARNm de COVID-19 codifican la proteína espiga del SARS-CoV-2;
- en las células vivas de los mamíferos, las instrucciones para construir una determinada molécula de proteína se almacenan como un gen dentro del ADN dentro del núcleo;
- para construir una molécula de proteína determinada, la célula primero transcribe su gen en ARN y modifica los dos extremos de esta molécula para formar ARN mensajero (ARNm). Luego, el ARNm se transporta desde el núcleo hasta el citoplasma, donde induce a las fábricas de proteínas de la célula, los ribosomas , a traducir la secuencia de nucleótidos del ARNm en la secuencia de aminoácidos correspondiente y ensamblar la proteína.
1.2. Pasos en la fabricación de vacunas de ARNm
Dado que la proteína espiga es una molécula grande, también lo es el ARNm que la codifica. La síntesis química total de grandes moléculas de ARNm no es práctica a escala. Por lo tanto, para obtener la molécula de ARNm que codifica la espiga, se imita in vitro el proceso mediante el cual las células producen sus propios ARNm. Esto implica los siguientes pasos:
- Una copia de ADN del gen de la proteína de punta se inserta en un plásmido bacteriano . Esta es una molécula de ADN de doble cadena en forma de anillo que puede existir en una célula bacteriana independientemente del propio ADN cromosómico de la célula, y que también puede copiarse y transmitirse a ambas células hijas cuando esa célula se divide.
- El plásmido recombinante (artificial) que porta el gen de la proteína espiga se introduce en una célula de la especie bacteriana Escherichia coli ( E. coli ). Desde E. colilas células se dividen muy rápidamente, esta célula puede crecer en poco tiempo hasta convertirse en un gran número de células. Cada una de estas células de la progenie contendrá sus propias copias heredadas del plásmido y, por lo tanto, del gen de la proteína espiga. Si bien existe una cierta posibilidad de que algunos de los descendientes pierdan el plásmido durante las sucesivas divisiones celulares, podemos hacer cumplir su mantenimiento mediante dándole un marcador seleccionable, que asegura que sólo sobrevivirán aquellas células que retengan el plásmido. Con los plásmidos utilizados tanto por Pfizer como por Moderna, este marcador de selección es un gen que dota a las células huésped de resistencia al antibiótico kanamicina. Para aplicar la selección, las bacterias simplemente se cultivan en presencia de kanamicina.
- Después de hacer crecer una cantidad suficiente de células bacterianas en un caldo nutritivo que contiene kanamicina, estas células se rompen y el ADN plasmídico se purifica de los otros componentes de las células bacterianas.
- Las moléculas de plásmido en forma de anillo se convierten en forma lineal utilizando una enzima de restricción , que escinde ambas hebras de la molécula de ADN en un sitio único y específico que se encuentra aguas abajo del gen de la proteína de pico. Este paso es necesario para evitar la formación de moléculas de ARN que son demasiado largas y pueden tener efectos no deseados in vivo. Las vacunas de Pfizer y Moderna no son de conocimiento público.
- Se usa una polimerasa de ARN , en presencia de los cofactores y los bloques de construcción de nucleósidos necesarios, para copiar el gen de la proteína espiga de la versión de ADN en el plásmido linealizado en la versión de ARNm. Tanto Pfizer como Moderna emplean la polimerasa de ARN T7, que se deriva del bacteriófago del mismo nombre. Esta enzima se une a una secuencia promotora análoga derivada igualmente de T7 que se ha modificado en el plásmido aguas arriba del gen de la proteína espiga. Esta interacción entre la polimerasa y el promotor inicia la transcripción. En esta etapa, el nucleósido sintético N-metil-pseudouridina(mψU) se incorpora al ARN artificial en lugar del nucleósido de uridina natural. Cuando se administra en forma de vacuna, el ARN modificado de esta manera estimula menos el sistema inmunitario innato que el ARN que contiene la uridina natural. También se traduce más eficientemente en proteína y, bajo ciertas condiciones, es más resistente a la degradación [ 1 ] . Tanto las vacunas de ARNm de Pfizer como las de Moderna contienen mψU en lugar de uridina.
- Los dos extremos de cada molécula de ARN se acoplan enzimáticamente a ciertos restos que también se encuentran en estas posiciones dentro de los ARNm de mamíferos naturales, y que mejoran su actividad biológica y estabilidad in vivo.
Estos pasos proporcionan un ARNm funcional que es capaz de instruir a los ribosomas de las células para que produzcan la proteína de punta. Sin embargo, en esta etapa el producto aún no es puro: todo el ADN molde derivado de bacterias todavía está presente. Este último no debe incluirse en el producto farmacéutico final, porque presenta riesgos para la salud de los destinatarios (consulte la Sección 4 ). Para deshacerse de este ADN, se agrega otra enzima llamada ADNasa . Esto debería romper el ADN en fragmentos más pequeños, que luego se pueden eliminar de las moléculas de ARN mucho más grandes mediante filtración y otras técnicas de purificación. En el paso final, el ARNm se combina con una mezcla de lípidos para empaquetarlo en nanopartículas lipídicas (LNP), que inducen a las células humanas a tomar la molécula de ARNm y producir la proteína de punta.
2. ¿Qué sabíamos previamente sobre el problema de la contaminación del ADN?
En pocas palabras, muy poco. Los informes de evaluación de la FDA sobre ambas vacunas [ 2 , 3 ] no mencionan el problema en absoluto. El informe de evaluación de la Agencia Europea de Medicamentos (EMA) sobre la vacuna de Pfizer menciona que “La solidez del paso de digestión con ADNasa no se considera demostrada de forma exhaustiva” [ 4 , p. 17] . Se utiliza un lenguaje similar en el informe de la EMA sobre la vacuna Moderna [ 5 , p. 19f] . Sin embargo, basándose únicamente en esta escasa información, es imposible decir si el problema se consideró grave y qué remedios exigió el regulador, en su caso.
3. Evidencia independiente sobre la contaminación por ADN de productos de ARNm
Al 3 de abril de 2023, Kevin McKernan describió sus hallazgos recientes en tres artículos en su sitio Substack [ 6 – 8 ]. Los experimentos descritos en los dos primeros informes se llevaron a cabo en muestras de vacunas “bivalentes” recientemente introducidas por Pfizer y Moderna. Estas preparaciones se asemejan a las anteriores “monovalentes” en su composición química, es decir, deben contener ARNm de alta pureza, complejado con una mezcla de moléculas lipídicas (similares a la grasa) en ARNm/nanopartículas lipídicas. La única diferencia entre las dos variedades es que las vacunas bivalentes contienen una mezcla de dos ARNm que codifican dos variantes antigénicas de la proteína espiga. Esto no tiene nada que ver con el problema técnico de la contaminación del ADN como tal. Sin embargo, observamos que el alcance de la contaminación del ADN puede variar entre los lotes de producción, y que hasta ahora solo se ha caracterizado un pequeño número de lotes a este respecto.
3.1. El primer informe de McKernan
En un estudio inicial [ 6 ] , McKernan caracterizó tanto el ARN como el ADN contenidos en las vacunas de ARNm.
3.1.1. Extracción y caracterización directa de ácidos nucleicos de las vacunas
El primer paso consistió en eliminar los lípidos para obtener los ácidos nucleicos puros. El método basado en solventes que utilizó no discrimina entre el ADN y el ARN: si ambos están presentes, ambos se recuperarán. Los ácidos nucleicos extraídos se separaron según el tamaño. Esto reveló no solo las especies de ARNm de punta regulares y de longitud completa esperadas, sino también fragmentos más pequeños, que habían sido observados previamente tanto por los reguladores como en el trabajo publicado por uno de los fabricantes [ 9 ] . Más sorprendentemente, también se encontraron especies de ARN más grandes que el ARNm de longitud completa. Estas especies permanecen sin caracterizar.
3.1.2. Amplificación de los ácidos nucleicos extraídos
Como paso preparatorio para determinar las secuencias de nucleótidos exactas de los ácidos nucleicos extraídos, se amplificaron mediante métodos de PCR. En el caso del ARN, la PCR fue precedida por la transcripción inversa en ADN usando una enzima dedicada (transcriptasa inversa). Dado que el objetivo principal de este estudio era estudiar el ARN en lugar del ADN, este paso de amplificación se sesgó contra el ADN mediante la adición de actinomicina D, que en las condiciones experimentales dadas inhibe selectivamente la síntesis de ADN. En consecuencia, se recuperaron cantidades relativamente bajas de ADN en la muestra amplificada. Sin embargo, en el caso de la vacuna de Pfizer, la cantidad de ADN que se determinó que estaba presente ya excedía el límite arbitrariamente decidido por la EMA para la proporción máxima permisible de ADN por ARN.
3.1.3. resultados de secuenciación de ADN
Tanto con los productos de Pfizer como con los de Moderna, se obtuvieron secuencias de ADN de plásmidos de ADN completos, aunque permaneció cierta ambigüedad en el caso de los plásmidos de Moderna. Por lo tanto, las características de las secuencias de plásmidos se discutirán en relación con el segundo estudio de McKernan, que usó cada vez más ADN puro para la secuenciación y, por lo tanto, proporcionó resultados más confiables.
3.2. Segundo informe de McKernan
El segundo estudio [ 7 ] se centró en cuantificar y caracterizar la contaminación de ADN que se detectó cualitativamente en el primero.
3.2.1. El ADN plásmido contenido en las vacunas de ARNm es competente para propagarse en células bacterianas
En el primer experimento, se determinó si el ADN plasmídico cuya presencia se había inferido a partir de los resultados de la secuenciación previa es efectivamente biológicamente funcional, en la medida en que pueda introducirse y persistir dentro de las células bacterianas. Con este fin, los ácidos nucleicos se extrajeron nuevamente de las muestras de vacunas. Estos ácidos nucleicos se mezclaron con una suspensión de células de E. coli que se habían vuelto competentes para la captación de ADN.
Después de inducir a estas células a tomar el ADN y darles algo de tiempo para que se recuperaran, se extendieron sobre placas de Petri llenas de medio de crecimiento solidificado que contenía kanamicina. Como se señaló anteriormente, la kanamicina matará cualquier célula de E. coli que no contenga un gen de resistencia a ella. Por lo tanto, el crecimiento observado de colonias bacterianas en esas placas de Petri confirmó que algunas células habían adquirido efectivamente resistencia a la kanamicina al absorber y propagar los plásmidos. Esto se observó con las muestras de vacunas de Pfizer y Moderna.
En este contexto, debemos señalar que solo las moléculas de plásmido circulares, pero no las linealizadas, pueden introducirse de manera eficiente en las células bacterianas. Por lo tanto, el éxito de este experimento sugiere que algunas de las moléculas del plásmido habían escapado al paso de linealización (paso 4 en la Sección 1.2).) y lo hizo durante todo el proceso de producción en la forma circular que existe en las células bacterianas. Por otro lado, dado que el número de colonias bacterianas observadas en este experimento no fue alto, es probable que la mayor parte del ADN se haya linealizado. Debido a que los peligros biológicos del ADN extraño dentro de nuestro propio cuerpo pueden variar dependiendo de si es lineal o circular, vale la pena señalar la presencia probable de ambas formas en las vacunas. Quedan por determinar las proporciones exactas de ADN circular y lineal en las mezclas.
3.2.2. La abundancia de ADN contaminante
El segundo hallazgo importante de este estudio es la cuantificación por PCR tanto del ADN como del ARNm contenidos en las muestras de vacunas. Como sabrá, en una reacción de PCR, un segmento elegido de una secuencia de ácido nucleico se duplica mediante síntesis enzimática en varios ciclos de reacción sucesivos. A partir del número de ciclos (o duplicaciones) necesarios para alcanzar un cierto umbral de concentración, podemos calcular cuántas copias de la secuencia objetivo estaban presentes al principio.
En estos experimentos, el formato experimental elegido fue PCR multiplex , es decir, se amplificaron dos secuencias diana en una sola mezcla de reacción. Uno de estos objetivos estaba dentro del gen de la proteína de punta y, por lo tanto, debería estar presente tanto en las moléculas de ADN del plásmido como en las moléculas de ARNm de punta transcritas a partir de ellas. Para incluir las moléculas de ARNm en esta amplificación, la PCR fue nuevamente precedida por la transcripción inversa.
La otra secuencia objetivo estaba dentro del gen de resistencia a la kanamicina, que debería estar presente solo en el ADN del plásmido. Al comparar el número de ciclos necesarios para que cada uno de los dos objetivos cruce el umbral, se determinó que hasta el 35 % del ácido nucleico total contenido en las vacunas es, de hecho, ADN. A modo de comparación, la EMA ha estipulado que el ADN no debe representar más del 0,033% del total de ácidos nucleicos.
3.2.3. Determinación de secuencias de ADN plasmídico
Los plásmidos que originalmente estaban contenidos en las vacunas y luego se introdujeron en las células bacterianas (consulte la Sección 3.2.1 ) se aislaron nuevamente de esos cultivos bacterianos y se determinaron sus secuencias de ADN completas. Dichas secuencias se proporcionaron en su totalidad en el primer estudio de McKernan [ 6 ] , pero indicó que todavía estaba trabajando para corroborar y refinar los datos de secuenciación. Mientras tanto, en la Figura 1 se muestran las características funcionales del ADN plasmídico encontrado en las muestras de vacunas de Pfizer . Se discutirán en relación con la evaluación de riesgos.
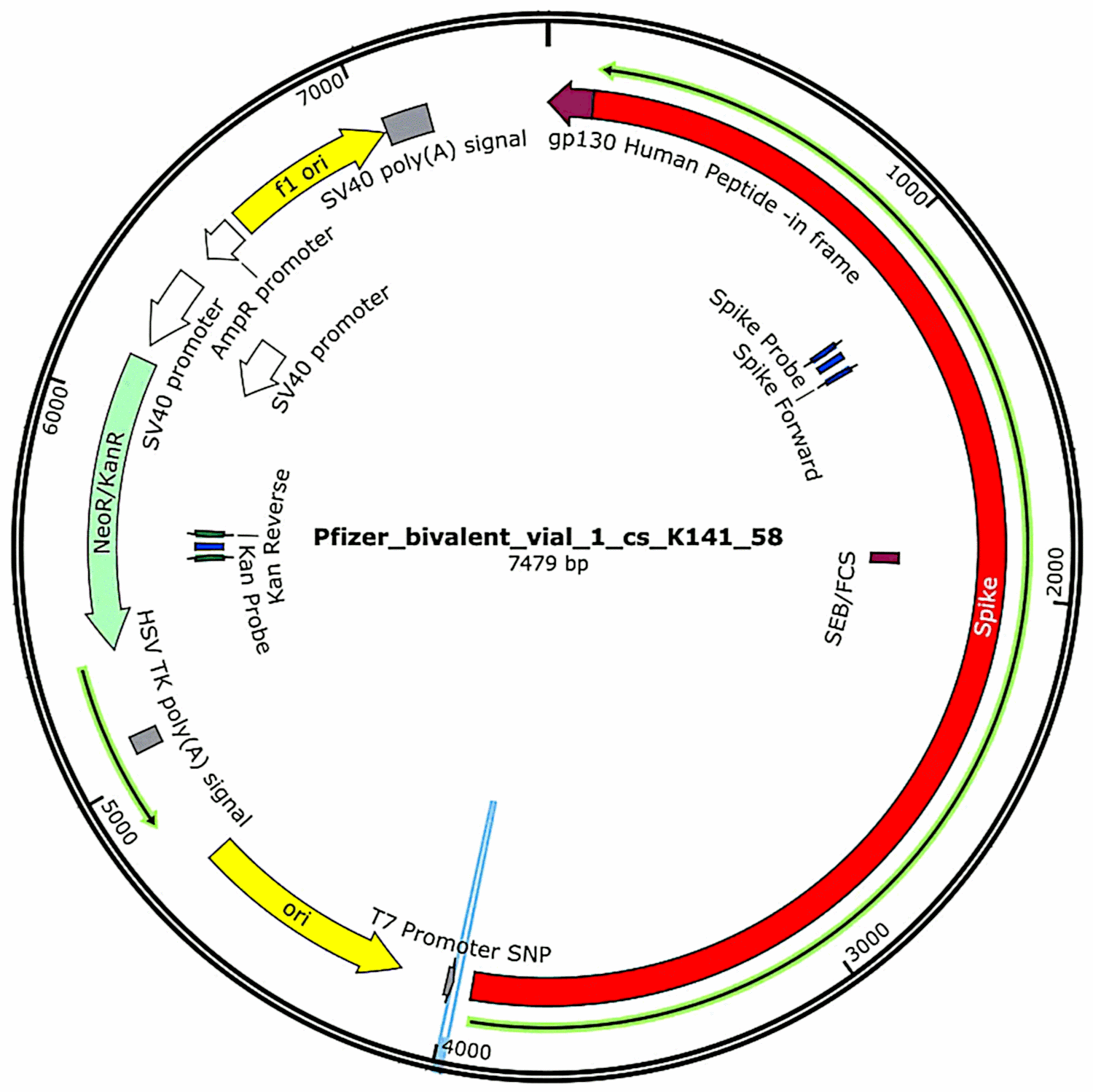
Figura 1:Mapa del ADN plasmídico contenido en uno de los viales de vacuna bivalente de Pfizer. Las características funcionales se deducen de la secuencia de ADN determinada experimentalmente. El gen que codifica la proteína espiga (rojo), cuya transcripción está impulsada por el promotor T7, representa aproximadamente la mitad de la secuencia total de ADN. El gen “NeoR/KanR” (verde claro) codifica una proteína que hará que las células bacterianas sean resistentes a la kanamicina o la neomicina, o que las células humanas sean resistentes al antibiótico relacionado G418. La secuencia amarilla etiquetada como “ori” es el origen bacteriano de la replicación; hará que se hagan copias del plásmido dentro de la célula bacteriana. Los elementos derivados de SV40 en la parte superior izquierda pueden inducir la expresión de resistencia a G418 en células humanas y también incluyen un origen de replicación que puede provocar la propagación del plásmido en células humanas. Están ausentes en los plásmidos de Moderna, que por lo demás son similares a los de Pfizer. Ver el texto para más detalles. Figura adaptada de[ 7 ] .
3.3. Tercer informe de McKernan
En su último informe hasta el momento, McKernan examinó ocho viales de un lote anterior de la vacuna de Pfizer utilizando el método de PCR cuantitativo descrito anteriormente. El contenido de ADN en este caso fue notablemente más bajo que con las muestras de vacunas bivalentes, pero aun así superó el límite de EMA por un factor de 18-70 [ 8 ] .
4. Evaluación de riesgos
Debemos suponer que el ADN recombinante que se encuentra en las vacunas de ARNm se puede introducir en las células de nuestro cuerpo, y que esto se verá favorecido por las nanopartículas lipídicas, al igual que ocurre con el propio ARNm. Esto plantea varios tipos diferentes de riesgos para la salud.
4.1. Duración extendida de la expresión de proteína de pico
Un argumento clave que se usa regularmente para promover la percepción de la seguridad de la vacuna de ARNm es que el ARNm es de corta duración in vivo y que, por lo tanto, la expresión del antígeno codificado también será de corta duración. Por ejemplo, el informe de evaluación de la EMA sobre los estados de la vacuna de Pfizer, con respecto a los experimentos con animales en una vacuna modelo que se aceptaron en lugar de los estudios adecuados sobre la vacuna COVID-19 real [4 , p . 46] :
Estos hallazgos parecen estar de acuerdo con dos estudios in vitro que compararon la duración de la expresión de proteínas entre especies de ARN mensajero, que eran idénticas en secuencia pero contenían uridina o mψU, respectivamente; como se señaló anteriormente, este último también está presente en las vacunas de ARNm de Pfizer y Moderna. En ambos estudios [ 1 , 10 ] , las especies de ARN modificadas con mψU indujeron niveles significativamente más altos de expresión de proteínas; sin embargo, esta expresión elevada disminuyó con una vida media similar a la del ARN no modificado. Ninguna de las vidas medias que pueden deducirse de los datos en cualquiera de los estudios supera los 4,5 días.
Sin embargo, está claro a partir de múltiples estudios en individuos vacunados que tanto la proteína espiga como los ácidos nucleicos que la codifican pueden detectarse, en el torrente sanguíneo y en varios órganos, durante semanas e incluso meses después de la inyección [ 11 – 15 ] . Esta discrepancia entre los estudios in vitro e in vivo hasta ahora ha sido difícil de entender. Los altos niveles de ADN plásmido residual en las vacunas detectadas por McKernan ahora sugieren una explicación plausible.
Para que el ADN del plásmido bacteriano admita la expresión prolongada de la proteína espiga, deben cumplirse dos condiciones:
- el ADN del plásmido debe persistir dentro de las células de nuestro cuerpo, y
- el gen de la proteína espiga en ese plásmido debe ser transcrito en ARNm por nuestra propia ARN polimerasa II celular.
Si bien aún no tenemos datos experimentales directos sobre los plásmidos de expresión de picos de Pfizer y Moderna, el precedente sugiere que, de hecho, se cumplen ambos requisitos. Se ha descubierto que los plásmidos recombinantes que expresan el factor IX de la coagulación persisten en las células hepáticas de los animales de experimentación a niveles estables durante un máximo de 1,5 años [ 16 , 17 ] , que fue la duración total del experimento. Se podría objetar que los plásmidos utilizados en estos estudios eran circulares, mientras que la mayor parte del ADN plasmídico contenido en las vacunas de ARNm probablemente se encuentra en forma lineal (consulte la Sección 1.2 ) . En respuesta, observamos que, en primer lugar, es probable que quede algo de ADN plásmido circular (consulte la Sección 3.2.1) .) y, en segundo lugar, que se ha demostrado que el ADN viral recombinante persiste en forma lineal dentro de los animales durante períodos de tiempo igualmente prolongados [ 18 ] , lo que sugiere que lo mismo puede ocurrir con el ADN plasmídico.
En los estudios citados [ 16 , 17 ] , el gen que codifica la proteína de interés (factor IX) había estado bajo el control de un promotor de mamíferos y, de hecho, la proteína del factor IX se expresó a niveles estables en todo momento. Por el contrario, el gen de la proteína espiga contenido en los plásmidos de expresión de Pfizer y Moderna está bajo el control de un promotor del bacteriófago T7. No podemos suponer a priori que este promotor funcionará en ausencia de su ARN polimerasa T7 afín. Sin embargo, se ha confirmado experimentalmente que, de hecho, el promotor T7 también se une a la ARN polimerasa II celular y provoca la expresión de proteínas en células de mamíferos [ 19 ] .
En resumen, la posibilidad de que la expresión de larga duración observada de la proteína espiga sea causada por el ADN plasmídico contenido en las vacunas de ARNm debe tomarse en serio. La persistencia prolongada del ARNm de la proteína espiga y su expresión después de la vacunación, detectada en biopsias y autopsias, se ha relacionado inequívocamente con un daño grave [ 14 , 20 ] , que probablemente esté mediado por un ataque inmunitario a las células que expresan este antígeno extraño. La omisión de los estudios experimentales correspondientes en la etapa de ensayos preclínicos, junto con la escala de esta contaminación, crea un riesgo de seguridad del todo inaceptable.
4.2. Riesgos asociados con las secuencias de ADN reguladoras derivadas de SV40
Una característica que identificó McKernan en los plásmidos de expresión de Pfizer pero no en los de Moderna [ 6 ] es un promotor derivado del virus SV40, que pertenece a la familia de los poliomas (consulte la Sección 4.2 ). Este promotor se encuentra aguas arriba del gen de resistencia a la kanamicina; y dado que es activo en células de mamíferos, la proteína codificada por este gen de resistencia se expresará en cualquier célula que contenga este ADN. Al igual que la proteína espiga, esta proteína es un antígeno extraño y, por lo tanto, también puede desencadenar un ataque inmunitario en las células que la expresan.
El promotor SV40 también incluye un origen interno de replicación que potencialmente puede hacer que se produzcan copias del plásmido dentro de las células de mamíferos [ 21 ] . Esto requerirá la presencia del antígeno T grande viral , una proteína que reconoce directamente este origen y luego inicia la replicación de la molécula de ADN. Esta proteína no está codificada por el plásmido, ni está normalmente presente en las células de nuestro cuerpo, pero podría ser suministrada por el propio virus SV40 o por un poliomavirus relacionado. Una minoría de la población humana está infectada de forma latente con SV40, y dicha infección latente está asociada con algunas enfermedades malignas y no malignas [ 22 ]. Si se toma una copia del plásmido de Pfizer en una célula que alberga SV40, es posible que se formen copias adicionales.
Dos poliomavirus relacionados que están mucho más extendidos en la población humana son el BK y el virus JC [ 23 , 24 ] . El antígeno T grande de JC aparentemente es menos eficaz junto con el origen de SV40 que la propia proteína de SV40 [ 25 ] , pero no se puede descartar la replicación del plásmido de Pfizer en células infectadas de forma latente con virus JC o BK. Copias adicionales del plásmido generado de esta manera amplificarían todos los otros riesgos discutidos en esta sección, con la posible excepción de inflamación no específica (consulte la Sección 4.4 ).
4.3. Inserción genómica del ADN plasmídico
Todos los escenarios discutidos hasta ahora involucran la persistencia episomal independiente del ADN del plásmido; estará presente cerca de los cromosomas (dentro del núcleo celular), pero no se habrá convertido en parte integral de ninguno de ellos. Estas moléculas de plásmido independientes y no replicantes tenderán a perderse durante la división celular [ 26 ] . Sin embargo, como veremos, en algunos casos una molécula de plásmido puede integrarse en uno de los cromosomas de su célula huésped y luego será heredada por todos los descendientes de esa célula.
La integración cromosómica es una forma de “genotoxicidad”, es decir, toxicidad que causa daño genético. Con respecto a la posibilidad de tales efectos, el informe de evaluación de la EMA sobre la vacuna de ARNm de Pfizer señala sucintamente [ 4 , p. 50] :
Aparentemente, los expertos de la EMA suponían que el ARN en general no afectaría la integridad del genoma de la célula huésped. Este punto de vista es incorrecto, y la primera evidencia para demostrarlo ha celebrado recientemente su quincuagésimo aniversario [ 27 ] . Sin embargo, la detección de copiosas cantidades de ADN plasmídico en las vacunas de ambos fabricantes ahora obvia la necesidad de presentar ese caso. Seguramente, incluso los científicos de la EMA sabrán que este ADN puede integrarse en el genoma de las células huésped humanas. No se necesitan características de secuencia específicas para que se produzca dicha integración y, en consecuencia, se ha observado de manera similar con el ADN de virus, bacteriófagos y plásmidos de mamíferos [ 28 ]. Vale la pena señalar que tales inserciones pueden ocurrir en ubicaciones aleatorias del genoma, pero los genes que se expresan activamente por la célula se ven afectados con mayor frecuencia [ 29 ] .
La integración cromosómica estable de un plásmido bacteriano en el ADN cromosómico de células de mamíferos se demostró ya en 1982 [ 30 ] . El plásmido en cuestión comparte múltiples características con los utilizados en la producción de vacunas de ARNm de Moderna y Pfizer. La introducción de genes extraños o modificados en células de mamífero utilizando esta y otras técnicas similares se ha convertido desde entonces en un lugar común en la investigación experimental y en la biotecnología. La metodología se denomina transfección y los organismos modificados de esta manera se denominan transgénicos . Observamos que la integración estable puede ocurrir tanto con ADN plásmido lineal como circular [ 31 ] .
En este contexto, también debemos considerar el estudio previamente publicado por Aldén et al. [ 32 ] , que detectó copias de ADN del gen de la proteína espiga en una línea de células hepáticas humanas después de que estas células hubieran estado expuestas a la vacuna de ARNm de Pfizer. Basándose en la suposición de que la vacuna contenía ARNm esencialmente puro pero no ADN, tomaron esta observación como evidencia de que el ARNm sintético había sufrido una transcripción inversa dentro de esas células. Su interpretación es plausible, porque se sabe que dicha transcripción inversa ocurre en principio, y se informó anteriormente en células de pacientes infectados con el virus SARS-CoV-2 [ 33 ]. Sin embargo, a la luz del descubrimiento de McKernan de que los viales de vacunas de Pfizer pueden contener cantidades sustanciales de ADN, parece igualmente posible que las observaciones de Aldén et al. indicaba simplemente la captación celular de este ADN. Sin embargo, de cualquier manera, sus hallazgos indican la presencia de ADN que codifica picos dentro de esas células, lo que indica un riesgo de inserción genómica.
4.3.1. Inserción genómica en terapia génica mediante vectores retrovirales
En una terapia génica adecuada, a menudo se desea la integración cromosómica, ya que corregirá el defecto genético en cuestión de forma duradera. Con este fin, se han desarrollado vectores de ADN especiales que tienen una propensión mucho mayor a sufrir tal integración. Estos vectores se derivan de retrovirus , cuya estrategia de supervivencia entera se basa en la integración genómica. Sin embargo, resulta que la integración, cuando ocurre en el lugar equivocado dentro del genoma, frecuentemente induce enfermedades malignas, especialmente leucemia [ 34 ] .. De hecho, esto es tan común que ha impedido la adopción generalizada de la terapia génica, incluso en enfermedades para las que todas las demás opciones terapéuticas también están cargadas de riesgos muy graves. Un buen ejemplo es la deficiencia de adenosina desaminasa, una enfermedad metabólica que acaba con los linfocitos y, por lo tanto, provoca una inmunodeficiencia combinada grave (SCID), una condición que sin tratamiento siempre es fatal durante la infancia. Esta enfermedad es, en principio, un objetivo muy adecuado para la terapia génica; sin embargo, un trasplante de médula ósea de un donante compatible y emparentado sigue siendo la opción terapéutica preferida, debido al grave riesgo de neoplasias malignas inducidas por la terapia génica [ 35 ] .
4.3.2. ¿Cómo la inserción genómica causa tumores malignos?
Nuestro genoma contiene múltiples genes que pueden dar lugar al cáncer si su nivel de expresión (la velocidad a la que se sintetizan las moléculas de ARNm y proteínas a partir de ellos) es demasiado bajo o demasiado alto. Es posible que una molécula de ADN extraño se inserte directamente en dicho gen y lo elimine por completo, o puede insertarse junto a él, y un promotor fuerte presente en ese ADN extraño puede causar una expresión excesiva del gen en cuestión. Además, se ha observado que los eventos de inserción también pueden causar cambios en la metilación del ADN en todo el genoma, lo que afectará los niveles de expresión de muchos genes; y algunos de estos cambios pueden contribuir a la inducción de malignidad. Es importante destacar que este efecto se ha observado no solo con el ADN viral, sino también con plásmidos bacterianos [ 36 ] .
Cuando las células se aíslan de un órgano humano o animal sano y se cultivan en cultivo celular, se dividirán durante un número limitado de generaciones y luego morirán. Por el contrario, las células derivadas de tumores malignos y leucemias pueden propagarse indefinidamente. También puede ocurrir un cambio similar en las células cultivadas, que por lo tanto se inmortalizan y, por lo general, también pierden algunas características que son características de su tejido de origen. Esta transformación puede inducirse, por ejemplo, infectando las células con el virus SV40 antes mencionado. De manera similar, las células se pueden transformar mediante transfección con un plásmido derivado de SV40 que retiene las partes cruciales del genoma viral, incluido el gen que codifica el antígeno T grande. Por otro lado, si falta el antígeno T grande en el plásmido, la transformación no suele ocurrir.[ 30 ] . Sin embargo, se han informado algunas excepciones [ 37 , 38 ] . Estos casos deben haber surgido de la alteración o desregulación de los genes celulares implicados en el control de la proliferación.
4.3.3. Integración genómica en células germinales
Los ovocitos se pueden transfectar in vivo en ciertas etapas de maduración [ 39 ] , al igual que las células productoras de esperma dentro de los testículos [ 40 ] . En este último caso, se demostró que las crías de los animales sometidos a dicho tratamiento eran transgénicas. Por lo tanto, no se puede descartar que las personas inyectadas con vacunas de ARNm que también contienen ADN den a luz posteriormente a niños transgénicos. La inserción de ADN en las células de la línea germinal también podría interferir con el desarrollo intrauterino temprano y, por lo tanto, inducir abortos espontáneos o malformaciones.
4.3.4. ¿Cómo debemos evaluar el riesgo de la inserción genómica?
Ciertamente es cierto que los plásmidos bacterianos tienen una menor propensión a insertarse en nuestro ADN cromosómico que los vectores de terapia génica especialmente diseñados para una integración eficaz. Pero, ¿cuán grande es exactamente el riesgo en el caso de los plásmidos contenidos en las vacunas de ARNm? La respuesta simple es que nadie lo sabe. Esto no se debe a que en principio sea incognoscible, sino a que no se realizaron los estudios experimentales apropiados en animales y, posteriormente, en humanos; o si lo fueran, los resultados se han ocultado al público, y aparentemente también a los reguladores.
¿Cómo se evaluarían tales riesgos en los procedimientos de aprobación llevados a cabo correctamente? La guía actual de la FDA sobre la prueba y aprobación de terapias génicas [ 41 ] recomienda que, en la etapa de prueba clínica, los pacientes sean monitoreados durante 15 años completos después de la administración, con exámenes anuales durante los primeros cinco años. Esto se aplica a los vectores con los que se pretende la inserción cromosómica. El documento de orientación continúa construyendo una falsa dicotomía entre vectores de inserción y no inserción, pero la línea divisoria entre ellos sigue siendo borrosa. Por un lado, la guía sugiere que
pero por otro lado dice que
La referencia citada en la última cita es un estudio de Wang et al. [ 42 ] , que identificaron inequívocamente la inserción de ADN de plásmido de ADN in vivo después de la inyección intramuscular, seguida de electroporación. Si bien la electroporación aumentó la absorción celular del ADN inyectado en relación con la inyección de ADN “desnudo”, probablemente fue mucho menos eficaz en este sentido que las nanopartículas lipídicas contenidas en las vacunas de ARNm. En consecuencia, debemos esperar cierto grado de integración cromosómica del ADN del plásmido contaminante in vivo.
4.4. Efecto proinflamatorio del ADN bacteriano
El sistema inmunológico innato humano reacciona con inflamación a varias macromoléculas bacterianas, incluido el ADN. Se debe suponer que las grandes cantidades de ADN presentes en las vacunas contribuyen a la inflamación cerca del lugar de la inyección y, potencialmente, también en otras partes del cuerpo.
5. Conclusión
La presencia de ADN plásmido contaminante en las vacunas de ARNm de Pfizer y Moderna conlleva graves riesgos para la salud, además de los ya conocidos y entendidos. Preeminente entre estos riesgos está la expresión prolongada de la proteína espiga, que puede conducir a una inflamación de tipo autoinmune correspondientemente prolongada y más destructiva, y la inducción de una enfermedad maligna después de la integración cromosómica del ADN del plásmido. Además, la magnitud de la contaminación demuestra de manera concluyente que los fabricantes no han dominado o implementado correctamente los procesos de producción diseñados. Cada uno de estos temas por sí solo sería motivo suficiente para exigir el retiro inmediato de estas vacunas.
Expresiones de gratitud
Agradecemos a Kevin McKernan y Ulrike Kämmerer por las correcciones y el debate.
Derechos de autor
Este texto tiene licencia bajo los términos de Creative Commons Attribution 4.0 International License (CC BY 4.0). Esto significa que eres libre de copiar y reutilizar el contenido, siempre que se acredite a los autores originales. Si realiza cambios en el texto, debe indicarlo explícitamente. Para obtener más información, consulte el sitio web de Creative Commons [ 43 ] .
Referencias
- (2015) El ARNm incorporado con N 1 -metilpseudouridina supera al ARNm incorporado con pseudouridina{}al proporcionar una expresión proteica mejorada y una inmunogenicidad reducida en líneas celulares de mamíferos y ratones . J.Control. Comunicado 217:337-344
- (2020) Documento informativo de la FDA: Pfizer-BioNTech COVID-19 Vaccine .
- (2020) Documento informativo de la FDA: Moderna MRNA-1273 .
- (2021) Informe de evaluación de la EMA: Comirnaty .
- (2021) Informe de evaluación de la EMA: COVID-19 Vaccine Moderna .
- (2023) La secuenciación profunda de las vacunas bivalentes de Moderna y Pfizer identifica la contaminación de los vectores de expresión diseñados para la amplificación de plásmidos en bacterias .
- (2023) Las vacunas bivalentes de Pfizer y Moderna contienen un 20-35 % de vector de expresión y son capaces de transformación en E.coli .
- (2023) Contaminación de ADN en 8 viales de vacunas de ARNm monovalente de Pfizer .
- (2023) Caracterización del ARNm de BNT162b2 para evaluar el riesgo de traducción de antígenos fuera del objetivo . J. Pharm. ciencia DOI:10.1016/j.xphs.2023.01.007
- (2018) Las vacunas de ARNm modificadas con nucleósidos inducen respuestas de células B del centro germinal y auxiliares foliculares T potentes . Exp. J. Medicina. 215:1571-1588
- (2021) Innovador: la vacunación con BNT162b2 (Pfizer-BioNTech) induce la vacunación con BNT162b2 (Pfizer-BioNTech) antes del desarrollo de anticuerpos: un mecanismo novedoso para la activación inmunitaria mediante vacunas de ARNm . J. Immunol. 207:2405-2410
- (2023) Las secuencias de la vacuna de ARN de pico de SARS-CoV-2 circulan en la sangre hasta 28 días después de la vacunación con COVID-19 . APMIS 131:128-132
- (2022) El ARNm de la vacuna se puede detectar en la sangre 15 días después de la vacunación . Biomedicinas 10:1538
- (2022) Caracterización clínica y molecular de un caso raro de miositis asociada a la vacuna COVID-19 mRNA BNT162b2 . vacunas 10
- (2022) Impresión inmunológica, amplitud del reconocimiento de variantes y respuesta del centro germinal en la infección y vacunación humana por SARS-CoV-2 . Celda DOI:10.1016/j.cell.2022.01.018
- (2001) Expresión génica hepática a largo plazo y de nivel terapéutico del factor IX humano después de la transferencia de plásmido desnudo in vivo . mol. El r. 3:947-57
- (2003) Corrección fenotípica completa y sostenida de la hemofilia B en ratones después de la transferencia de genes hepáticos de un plásmido de factor IX humano de alta expresión . J. Trombo. Hemost. 1:103-11
- (2009) Persistencia de vectores adenovirales de alta capacidad como genomas monoméricos defectuosos en replicación in vitro y en hígado murino . Tararear. Gene Ther. 20:883-96
- (2000) [La función del promotor T7 como elementos que actúan en cis para la polimerasa II en células eucariotas] . Yi Chuan Xue Bao 27:455-61
- (2022) Informe de un caso: encefalitis necrosante multifocal y miocarditis después de la vacunación con ARNm BNT162b2 contra Covid-19 . Vacunas 10:2022060308
- (1983) Definición de la región promotora temprana del virus del simio 40 y demostración de un sesgo de rango de huéspedes en el efecto de potenciación de la repetición de 72 pares de bases del virus del simio 40 . proc. nacional Academia ciencia Estados Unidos 80:721-5
- (2019) Asociación entre el virus simio 40 y tumores humanos . Frente. oncol. 9:670
- (2013) Una cornucopia de poliomavirus humanos . Nat. Rev. Microbiol. 11:264-76
- (2020) Poliomavirus humanos BK y JC: conocimientos moleculares y prevalencia en Asia . Resolución de virus 278:197860
- (1985) Replicación diferencial de SV40 y ADN de polioma en células de ovario de hámster chino . Resolución de virus 2:329-44
- (2003) Persistencia episomal de genomas de vectores adenovirales recombinantes durante el ciclo celular in vivo . J.Virol. 77:7689-95
- (1970) ADN polimerasa dependiente de ARN en viriones de virus tumorales de ARN . Naturaleza 226:1209-11
- (2016) Cuidado con las manipulaciones en el genoma: desestabilización epigenética a través de inserciones de ADN (extranjero) . Epigenómica 8:587-91
- (1996) Un nuevo concepto en oncogénesis (adenoviral): integración de ADN extraño y sus consecuencias . bioquimica Biografía. Acta 1288:F79-99
- (1982) Transformación de células de mamífero a resistencia a antibióticos con un gen bacteriano bajo el control del promotor de la región temprana de SV40 . J. Mol. aplicación Gineta. 1:327-41
- (2010) Optimización de la generación de líneas celulares neuronales estables mediante la digestión con enzimas de restricción previa a la transfección del ADN plasmídico . Citotecnología 62:189-94
- (2022) Transcripción inversa intracelular de la vacuna de ARNm de Pfizer BioNTech COVID-19 BNT162b2 in vitro en línea celular de hígado humano . actual Cuestiones Mol. Biol. 44:1115-1126
- (2020) ARN del SARS-CoV-2 transcrito de forma inversa e integrado en el genoma humano . bioRxiv DOI:10.1101/2020.12.12.422516
- (2008) Sola dosis facit venenum. Leucemia en ensayos de terapia génica: ¿una cuestión de vectores, insertos y dosis? . Leucemia 22:1849-1852
- (2017) Cómo gestionamos la inmunodeficiencia combinada grave con deficiencia de adenosina desaminasa (ADA SCID) . J. Clin. inmunol. -:n / A
- (2018) Respuesta epigenética hereditaria hacia la entrada de ADN extraño por células huésped de mamíferos: un guardián de la estabilidad genómica . Epigenética 13:1141-1153
- (1995) Transformación de alta eficiencia de células endoteliales humanas mediante transfección mediada por Apo E con ADN plasmídico . Bioquímica Biografía. Res. común 214:206-11
- (2002) Transformación de células MC3T3-E1 después de estrés y transfección con plásmido pSV2neo . Res. contra el cáncer. 22:585-98
- (2003) Transfección de ovocitos y otros tipos de células ováricas en conejos después de la inyección directa en las arterias uterinas de adenovirus y plásmidos/liposomas . Gene Ther. 10:580-4
- (2008) Transgénesis a través de la integración permanente de genes en la repoblación de células espermatogoniales in vivo . Nat. Métodos 5:601-3
- (2020) Seguimiento a largo plazo después de la administración de productos de terapia génica humana: orientación para la industria .
- (2004) Detección de la integración del ADN plasmídico en el ADN genómico del huésped después de la inyección intramuscular y la electroporación . Gene Ther. 11:711-21
- (2023) Creative Commons Attribution 4.0 International License (CC BY 4.0) .
Este artículo fue publicado originalmente en https://doctors4covidethics.org/. Lea el original.
Periodistas por la Verdad no realiza valoraciones sobre los estudios de terceros. Para consultar algunas versiones alternativas les recomendamos leer: Addressing misinformation about excessive DNA in the mRNA vaccines | Therapeutic Goods Administration (TGA)

 VERDADERO
VERDADERO FALSO
FALSO