AUTORES
Feria Hikmet, Research engineer at Department of Immunology, Genetics and Pathology, Clinical and experimental pathology; HPA Production Cecilia Lindskog Bergström, Sweden (Suecia), Loren Méar, Department of Immunology, Genetics and Pathology, Clinical and experimental pathology; HPA Production Cecilia Lindskog Bergström, Sweden, Mathias Uhlén, KTH School of Engineering Sciences in Chemistry, Biotechnology and Health, Department of Protein Science Division of Systems Biology AlbaNova University Center, Sweden, Cecilia Lindskog, Department of Immunology, Genetics and Pathology Uppsala University | UU , Sweden
El proyecto fue financiado por la Fundación Knut y Alice Wallenberg. Los patólogos y el personal del Departamento de Patología Clínica del Hospital Universitario de Uppsala son reconocidos por proporcionar los tejidos utilizados para la secuenciación de ARN y la inmunohistoquímica. Los autores también desean agradecer a todo el personal del Atlas de proteínas humanas por su trabajo.
Artículo publicado en Molecular Systems Biology (doi: 10.15252 / msb.20209610)
El nuevo SARS-coronavirus 2 (SARS-CoV-2) plantea un desafío global para la salud y la sociedad. Para comprender la susceptibilidad a la infección por SARS-CoV-2, es necesaria la expresión específica del tipo de célula del receptor de la superficie de la célula huésped. La proteína clave que se sugiere que está involucrada en la entrada de la célula huésped es la enzima convertidora de angiotensina I 2 (ACE2). Aquí, informamos el patrón de expresión de ACE2 en> 150 tipos de células diferentes correspondientes a todos los principales tejidos y órganos humanos basados en un riguroso análisis inmunohistoquímico. Los resultados se compararon con varios conjuntos de datos tanto a nivel de ARNm como de proteína. La expresión de ACE2 se observó principalmente en enterocitos, túbulos renales, vesícula biliar, cardiomiocitos, células reproductoras masculinas, trofoblastos placentarios, células ductales, ojos y vasculatura. En el sistema respiratorio, la expresión fue limitada, con una expresión baja o nula en un subconjunto de células en unos pocos individuos, observada por un solo anticuerpo. Nuestros datos constituyen un recurso importante para futuros estudios sobre la entrada de células hospedadoras del SARS-CoV-2, con el fin de comprender la biología de la enfermedad y ayudar en el desarrollo de tratamientos efectivos para la infección viral.
Tal y como se muestra en este meticuloso estudio, cuyos resultados podemos corroborar en The Human Protein Atlas, la proteína ACE2 se manifiesta principalmente en las células de los riñones y el sistema reproductivo. En el sistema respiratorio la expresión es limitada, y en un subconjunto de células de individuos no había ninguna expresión o sólo una expresión baja en el sistema respiratorio.
RESUMEN
Para comprender la susceptibilidad a la infección por SARS-CoV-2, es necesaria la expresión específica del tipo celular del receptor de la superficie de la célula huésped. La proteína clave que se sugiere que está involucrada en la entrada de la célula huésped es la enzima convertidora de angiotensina I 2 (ACE2), y su expresión se ha informado en varios órganos humanos, en algunos casos con resultados inconsistentes o contradictorios. Aquí, nuestro objetivo es verificar un perfil de expresión confiable de ACE2 en todos los principales tipos de células y tejidos humanos. Con base en análisis inmunohistoquímicos estrictamente validados y secuenciación de ARNm de alto rendimiento de varios conjuntos de datos, encontramos que la expresión de ACE2 se localiza principalmente en microvellosidades del tracto intestinal y túbulos proximales renales, epitelio de la vesícula biliar, células de Sertoli testiculares y células de Leydig, células glandulares de la vesícula seminal y cardiomiocitos. No se pudo confirmar la expresión en varias otras ubicaciones informadas anteriormente, incluidas las células alveolares de tipo II (AT2). Además, la expresión de ACE2 estuvo ausente en la línea celular de carcinoma de pulmón AT2 A549, que se usa a menudo como modelo para estudios de replicación viral. Nuestro análisis sugiere que la expresión de ACE2 en el sistema respiratorio humano parece ser limitada, y la expresión del receptor en el epitelio pulmonar o respiratorio a nivel de proteínas aún no se ha confirmado.





INFORME ORIGINAL DE LOS AUTORES
OBJETO
Para una comprensión completa de la susceptibilidad a la infección por SARS-CoV-2 y el papel de ACE2, es necesario estudiar la expresión específica del tipo celular de ACE2 en tejidos humanos, tanto a nivel de ARNm como de proteínas. En la presente investigación, proporcionamos un resumen completo de los niveles de expresión de ACE2 en todos los principales tejidos humanos basados en múltiples estudios previos, tanto a nivel de ARNm como de proteína. Confirmamos los hallazgos con la localización específica del tipo celular de ACE2 en 78 tipos diferentes de células humanas
INTRODUCCIÓN
Los coronavirus son viriones de ARN envueltos, y varios miembros de la familia Coronaviridae pertenecen a las causas más frecuentes de resfriado común. Dos coronavirus anteriores que se transmitieron de animales a humanos han causado una enfermedad grave; el coronavirus del síndrome respiratorio agudo severo (SARS-CoV) y el coronavirus del síndrome respiratorio de Oriente Medio (MERS- CoV). El nuevo coronavirus SARS-coronavirus 2 (SARS-CoV-2), que comparte aproximadamente el 80% de identidad de aminoácidos con el SARS-CoV, es el agente de la enfermedad por coronavirus 19 (COVID-19), la nueva pandemia de rápida propagación.
Cuando los coronavirus entran en la célula diana, una unidad de superficie de la glicoproteína de pico (S) se une a un receptor celular. Al entrar, las proteasas celulares escinden la proteína S, lo que conduce a la fusión de las membranas viral y celular. Se ha demostrado previamente que el SARS-Cov ingresa a la célula a través del receptor ACE2, preparado por la serina proteasa celular TMPRSS2, y estudios recientes sugieren que también el SARS-CoV-2 emplea ACE2 y TMPRSS2 para la entrada 1 de la célula huésped . La
Para una comprensión completa de la susceptibilidad a la infección por SARS-CoV-2 y el papel de ACE2, es necesario estudiar la expresión específica del tipo celular de ACE2 en tejidos humanos, tanto a nivel de ARNm como de proteínas. El sistema respiratorio es de especial interés debido a su alta susceptibilidad a los virus inhalados, y la mayoría de los virus que infectan a los seres humanos utilizan diferentes células epiteliales de las vías respiratorias para la replicación. La expresión de ACE2 en células alveolares de tipo II (AT2) sugeriría por tanto que estas células pueden servir como reservorio para la invasión viral y facilitar la replicación del SARS-CoV-2 en el pulmón. Sin embargo, también es importante estudiar otras ubicaciones de tejido que podrían servir como entrada potencial.
En la presente investigación, proporcionamos un resumen completo de los niveles de expresión de ACE2 en todos los principales tejidos humanos basados en múltiples estudios previos, tanto a nivel de ARNm como de proteína. Confirmamos los hallazgos con la localización específica del tipo celular de ACE2 en 78 tipos diferentes de células humanas basados en inmunohistoquímica utilizando dos anticuerpos independientes validados rigurosamente.
RESULTADOS
Expresión de ACE2 en tejidos humanos mediante perfiles de transcripción
La ACE2 es una carboxipeptidasa que regula negativamente el sistema renina-angiotensina (RAS), que puede inducir vasodilatación al escindir la angiotensina II. La proteína fue identificada en 2000 como un homólogo de la ECA 3 y debido a la importancia de la ECA en la enfermedad cardiovascular y al uso de inhibidores de la ECA para el tratamiento de la hipertensión arterial y la insuficiencia cardíaca, ha habido un gran interés en comprender la función y expresión de ACE2 en varios órganos humanos. En la Figura 1 , se muestra un resumen de los niveles de expresión en una selección de diferentes órganos, basado en conjuntos de datos disponibles públicamente en niveles tanto transcriptómicos como proteómicos.
Figura 1.
Un resumen de la expresión de ACE2 en tejidos humanos basado en conjuntos de datos de transcriptómica y proteómica disponibles públicamente.
Tres tamaños diferentes de círculos representan niveles de expresión altos, medios o bajos, y cada círculo está codificado por colores según el sistema de órganos. Se observó una expresión consistente en el tracto intestinal, la vesícula biliar, el riñón, los testículos y el músculo cardíaco en todos los conjuntos de datos. El perfil de expresión más amplio fue informado por Hamming
Con la introducción del análisis de microarrays, se lanzaron mapas de todo el cuerpo que cuantifican los patrones de expresión génica global basados en transcripciones de ARNm, como BioGPS 5 , pronto seguido de varios esfuerzos de transcriptómica a gran escala basados en la secuenciación de próxima generación en muestras que representan la mayoría de los órganos humanos importantes. y tejidos. Tres de estas iniciativas recientes incluyen el consorcio Human Protein Atlas (HPA) 6y el consorcio de expresión tisular basada en el genoma (GTEx) 7 basado en RNA-seq, y el consorcio FANTOM5 basado en la expresión génica del análisis cap (CAGE) 8 . El consorcio HPA tiene como objetivo caracterizar todo el proteoma humano en tejidos, órganos, células y orgánulos mediante una combinación de transcriptómica y proteómica basada en anticuerpos. En la base de datos de acceso abierto www.proteinatlas.org, se muestran perfiles de expresión basados en conjuntos de datos tanto externos como internos, junto con datos primarios, criterios utilizados para la validación e imágenes originales de alta resolución de muestras de tejido humano teñidas inmunohistoquímicamente. La HPA muestra datos de expresión de ARNm de HPA, GTEx y FANTOM5, y también resume los tres conjuntos de datos como niveles de expresión normalizados (NX) en 37 tejidos y órganos que cubren todas las partes principales del cuerpo humano, incluida la sangre humana 9 , 10 . El consenso de estos tres conjuntos de datos muestra niveles de expresión medios o altos en riñón, testículo y tracto intestinal, junto con baja expresión en el corazón y tejido adiposo y falta de expresión en la mayoría de los demás órganos normales, incluido el pulmón ( Figura 1 .
Expresión de ACE2 en tejidos humanos mediante perfiles basados en anticuerpos
Para estudiar el papel funcional de ACE2 en tejidos humanos, es necesario analizar la expresión a un nivel específico del tipo de célula, ya que subconjuntos más pequeños de células pueden expresar el receptor que puede estar por debajo del límite de detección cuando se mezcla con todas las demás células en una muestra de tejido compleja. Complementar los datos de transcriptómica con inmunohistoquímica no solo brinda la posibilidad de definir la localización de proteínas en diferentes compartimentos a nivel de una sola célula, sino que también proporciona información espacial importante en el contexto de células vecinas en muestras de tejido intacto. Además, la combinación de transcriptómica con proteómica proporciona una estrategia ventajosa para estudiar la representación funcional del genoma humano. Un estudio de inmunohistoquímica previo de Hamming et al 11
El análisis de ACE2 en el sistema respiratorio, la mucosa oral y la piel.
Si bien esto es en parte consistente con los datos de transcriptómica de experimentos de microarrays 5, los hallazgos son contradictorios con los tres conjuntos de datos de expresión de ARNm más recientes. En el sistema respiratorio, tanto las células epiteliales alveolares AT1 y AT2 como las células epiteliales de la nasofaringe se tiñeron claramente. Los autores también presentaron una expresión abundante y consistente de células endoteliales, células de músculo liso, fibroblastos y adipocitos de todos los tejidos examinados, a pesar de que varios de los órganos correspondientes que normalmente contienen fracciones altas de tales células estromales y mesenquimales tenían niveles de ARNm bajos o ausentes. . El análisis inmunohistoquímico de Hamming et al se basó en un solo anticuerpo, la calidad se aseguró mediante tres criterios diferentes: 1) incubación de las secciones de control con soluciones de anticuerpos anti-ACE2 preincubadas con el péptido sintético contra el que se generó el anticuerpo, 2) incubación de secciones de control con anticuerpos policlonales de conejo no relacionados, y 3) incubación con PBS en lugar de anticuerpo primario. Las tres secciones de control fueron negativas para la inmunohistoquímica; sin embargo, estos métodos de validación aún no prueban que el anticuerpo sea específico para ACE2 endógeno en muestras de tejido fijadas con formalina e incluidas en parafina. Por lo tanto, el anticuerpo no cumple con los criterios de validación mejorada propuestos por el Grupo de Trabajo Internacional para la Validación de Anticuerpos (IWGAV), formado con representantes de varias instituciones académicas importantes.
IWGAV propuso cinco pilares diferentes que se utilizarán para la validación de anticuerpos para garantizar la reproducibilidad de los estudios basados en anticuerpos y promover estrategias estrictas para la validación. Para garantizar que un anticuerpo se una a la proteína diana deseada, es necesario que la validación se haya realizado de una manera específica de la aplicación, utilizando al menos una de las estrategias sugeridas por IWGAV.
De acuerdo con las recomendaciones de IWGAV, la HPA ha introducido e implementado este concepto para mejorar la validación de los anticuerpos usando un método ortogonal. Además, la localización específica del tipo celular de la expresión de la proteína en 78 tipos de células diferentes fue similar entre los dos anticuerpos independientes. De los 78 tipos de células evaluados, solo se observó expresión confiable en 10 tipos de células: microvellosidades del duodeno, intestino delgado, colon y recto, túbulos renales proximales, en el epitelio de la vesícula biliar, células de Sertoli testiculares y células de Leydig, un subconjunto de células glandulares en vesícula seminal y en cardiomiocitos. (Figura 2). La tinción de los cardiomiocitos fue más difusa y principalmente citoplásmica, mientras que todas las demás ubicaciones positivas fueron, como se esperaba, más prominentes hacia la membrana plasmática. Un anticuerpo también mostró positividad en un subconjunto de membranas apicales en los conductos biliares del hígado, mientras que el otro anticuerpo fue consistentemente negativo en los conductos biliares de los tres individuos. Debido a la discrepancia entre los anticuerpos, la tinción en los conductos biliares para uno de los anticuerpos debe interpretarse con precaución, ya que puede representar una unión fuera del objetivo. No se pudo encontrar una expresión distinta confirmada por ambos anticuerpos en ninguno de los otros tipos de células analizados, incluidas las células epiteliales del sistema respiratorio, las células endoteliales de los vasos sanguíneos, las células del músculo liso y los fibroblastos.11 , y el presente perfil de expresión de todo el cuerpo muestra una alta correlación con diferentes conjuntos de datos de expresión de ARNm.
Figura 2.
Localización específica de tipo celular de ACE2 en tejidos humanos basada en inmunohistoquímica.
Imágenes representativas de 18 tipos de tejido y estructuras histológicas teñidas con inmunohistoquímica utilizando el anticuerpo policlonal de conejo HPA000288 (Atlas Antibodies AB), dirigido a la proteína ACE2 humana (marrón) y contrateñido con hematoxilina (azul). La expresión más alta de ACE2 se observó en microvellosidades del tracto intestinal y túbulos renales proximales, en membranas del epitelio de la vesícula biliar, células de Sertoli testiculares y células de Leydig, un subconjunto de células glandulares en la vesícula seminal (puntas de flecha) y en cardiomiocitos del citoplasma. Tenga en cuenta que la expresión de la proteína ACE2 no se detectó en las partes inferiores de la capa intestinal de la mucosa.
Otro método para determinar los niveles de proteína en una muestra de tejido es la espectrometría de masas, y en uno de los estudios a gran escala basados en espectrometría de masas de diferentes tejidos humanos normales 14 , 15 , se incluyeron muestras de pulmón. El conjunto de datos en este mapa de proteoma humano 14 generalmente se correlacionó bien con los niveles de expresión de ARNm de HPA, GTEx y FANTOM5, y los datos de IHC de HPA, mostrando la expresión más alta en riñón y testículo, y sin expresión en pulmón ( Figura 1 ).
Expresión de ACE2 en tejidos humanos utilizando perfiles de transcriptómica de células individuales
Recientemente, se han publicado varios conjuntos de datos que presentan la expresión de ACE2 en pulmones humanos basados en RNA-seq de una sola célula (scRNA-seq). Esta nueva plataforma para el análisis de células individuales constituye un complemento importante para la localización in situ por inmunohistoquímica y, por lo tanto, proporciona una herramienta excelente en los estudios de los niveles de transcripción que se espera encontrar en subconjuntos más pequeños de células en muestras de tejido complejas.
Sin embargo, los datos para ACE2 de múltiples conjuntos de datos de scRNA-seq en el tracto respiratorio humano no son concluyentes. En la Figura 3 , se proporciona un resumen de la expresión de ACE2 en el pulmón humano basado en tres conjuntos de datos de scRNA-seq diferentes disponibles al público 16 – 18con la expresión transcriptómica en células individuales de ACE2 y la proteína tensioactiva SFTPA1. Los datos de los tres conjuntos de datos revelan que solo una pequeña fracción de las células en el pulmón expresan ACE2 en un nivel bajo, en contraste con la abundante expresión de la proteína tensioactiva en grupos que representan células AT2. Esto contrasta con otros estudios que sugieren un enriquecimiento en las células AT2, aunque debe tenerse en cuenta que la expresión a menudo se identificó solo en fracciones muy pequeñas de las células AT2, a menudo menos del 1% 19 , 20 . También hay otros estudios que concluyen que la expresión de ACE2 es considerablemente mayor en otras células del tracto respiratorio, como las células ciliadas o secretoras 21 , 22., o en células epiteliales escamosas de mucosa oral o esófago 23 , 24 . De manera similar, en Human Cell Landscape (HCL) 16 publicado recientemente , los niveles de expresión de ACE2 fueron más altos en los tipos de células que respaldan los datos de la inmunohistoquímica basada en anticuerpos del programa HPA, incluidos los enterocitos yeyunales y duodenales, las células epiteliales de la vesícula biliar y los túbulos proximales renales. y células de Sertoli testiculares. En las tres muestras de pulmón adulto, el nivel de expresión fue bajo y no se observó expresión elevada en células AT2
Figura 3.
Análisis de secuencia de ARN unicelular de pulmón humano adulto.
Análisis de proyección y aproximación múltiple uniforme (UMAP) de células pulmonares basado en tres conjuntos de datos disponibles públicamente. Cada punto representa una sola celda coloreada de acuerdo con su identidad de grupo. No se pudo identificar una alta expresión específica de grupo de ACE2 en ninguno de los conjuntos de datos. El patrón de expresión del marcador de células AT2 SFTPA1 se muestra como referencia.
Expresión de ACE2 en líneas celulares humanas
Los modelos experimentales más comúnmente usados para evaluar el tropismo y la replicación de virus en humanos incluyen cultivos ex vivo de tejidos respiratorios humanos, células primarias del sistema respiratorio humano o líneas celulares derivadas del sistema respiratorio humano. La línea celular más utilizada en tales estudios es A549, derivada de un carcinoma de pulmón de células AT2
pulmón humano, incluida A549, la línea celular epitelial bronquial inmortalizada HBEC-KT y la línea celular de carcinoma de pulmón microcítico SCLC-21H. Los niveles de expresión también estaban por debajo del límite en la mayoría de las otras líneas celulares, sin embargo, se encontró una expresión baja (<5 NX) en BEWO (coriocarcinoma metastásico), HaCaT (queratinocitos), Hep G2 (carcinoma hepatocelular), NB-4 (leucemia promielocítica aguda). ), RPMI-8226 (mieloma múltiple), RT4 (vejiga urinaria) y SH-SY5Y (neuroblastoma). Esto sugiere que ninguna de las líneas de células pulmonares es un modelo apropiado para estudiar el papel de ACE2 en el sistema respiratorio humano. En el estudio de inmunohistoquímica anterior de Hamming et al, la positividad de ACE2 en células de A549 se presentó como una confirmación de expresión en células AT2. Debido a la falta de expresión de ACE2 en A549 en el nivel de ARNm y al hecho de que el mismo estudio sugirió expresión en múltiples otras localizaciones de tejido, ni confirmada por niveles de ARNm o por anticuerpos bien validados en la presente investigación, es probable que el La positividad previamente publicada de ACE2 en A549 se debió a la unión fuera del objetivo.
DISCUSIÓN
En la presente investigación, utilizamos dos anticuerpos independientes bien caracterizados para el análisis inmunohistoquímico de ACE2. Sobre la base de estrictos criterios de validación y la comparación de los perfiles de expresión de proteínas con múltiples conjuntos de datos transcriptómicos, la expresión confiable solo pudo confirmarse en microvellosidades del tracto intestinal y túbulos renales proximales, en el epitelio de la vesícula biliar, células de Sertoli testiculares y células de Leydig, un subconjunto de células glandulares en vesícula seminal y en cardiomiocitos, sin expresión detectable en epitelio pulmonar o respiratorio. No hay datos fiables que confirmen la expresión a nivel de proteína de ACE2 en células AT2, que anteriormente se ha demostrado que albergan ACE2. Expresión limitada de ACE2 en células AT2 sugerida en conjuntos de datos recientes basados en scRNA-seq, sin embargo, este hallazgo solo se observa en algunos de los individuos,
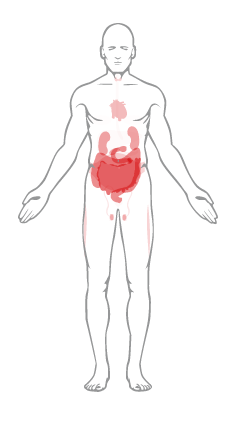
Todos los conjuntos de datos estudiados confirman una alta expresión constante en el tracto intestinal, la vesícula biliar y el riñón. Se necesitan más estudios para explorar la posible participación de la ECA2 en la manifestación de síntomas relacionados con estos niveles de expresión más altos de ACE2. Curiosamente, en un estudio reciente en individuos pediátricos con COVID-19, ocho de cada 10 casos mostraron hisopos rectales positivos para SARS-CoV-2, lo que sugiere que el tracto gastrointestinal puede diseminar el virus y que la transmisión fecal-oral puede ser una posible vía de infección.27 . También debe tenerse en cuenta que los requisitos previos de la interacción entre el patrón de unión del SARS-CoV-2 a los receptores de la superficie celular humana y la progresión de la enfermedad COVID-19 son multifactoriales, que incluyen no solo la unión del virus a las células diana, sino también otros factores. como el sistema inmunológico del huésped y la carga total de virus.
En resumen, sobre la base de múltiples conjuntos de datos sobre el nivel de ARNm y proteínas, utilizando métodos de células individuales y extractos de tejido más grandes, aquí mostramos que la expresión de ACE2 en tejidos humanos se localiza principalmente en las microvellosidades del intestino y los túbulos proximales renales, el epitelio de la vesícula biliar, Sertoli testicular células y células de Leydig, células glandulares de vesícula seminal y cardiomiocitos. Varias ubicaciones adicionales sugeridas por estudios anteriores, incluida la expresión en células AT2, no pudieron confirmarse. Esto plantea preguntas sobre el papel de la ECA2 para la infección de los pulmones humanos y destaca la necesidad de explorar más a fondo la ruta de transmisión durante la infección por SARS-CoV-2 para comprender la biología de la enfermedad y ayudar en el desarrollo de tratamientos efectivos para el paciente. infección viral.
MÉTODOS
Fuentes de datos
Footnotes
REFERENCES
[1].↵Hoffmann, M., Kleine-Weber, H., Schroeder, S., Kruger, N., Herrler, T., Erichsen, S., Schiergens, T. S., Herrler, G., Wu, N. H., Nitsche, A., Muller, M. A., Drosten, C., and Pohlmann,
S. (2020) SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor, Cell . Google Scholar
[2].↵Xu, X., Chen, P., Wang, J., Feng, J., Zhou, H., Li, X., Zhong, W., and Hao, P. (2020) Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike
[4].↵Harmer, D., Gilbert, M., Borman, R., and Clark, K. L. (2002) Quantitative mRNA expression profiling of ACE 2, a novel homologue of angiotensin converting enzyme, FEBS Lett 532, 107–110. CrossRef PubMed Web of Science Google Scholar
[5].↵Wu, C., Orozco, C., Boyer, J., Leglise, M., Goodale, J., Batalov, S., Hodge, C. L., Haase, J., Janes, J., Huss, J. W., 3rd., and Su, A. I. (2009) BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources, Genome Biol 10, R130. CrossRef PubMed Google Scholar
[6].↵Uhlen, M., Fagerberg, L., Hallstrom, B. M., Lindskog, C., Oksvold, P., Mardinoglu, A., Sivertsson, A., Kampf, C., Sjostedt, E., Asplund, A., Olsson, I., Edlund, K., Lundberg, E., Navani, S., Szigyarto, C. A., Odeberg, J., Djureinovic, D., Takanen, J. O., Hober, S., Alm, T., Edqvist, P. H., Berling, H., Tegel, H., Mulder, J., Rockberg, J., Nilsson, P., Schwenk, J. M., Hamsten, M., von Feilitzen, K., Forsberg, M., Persson, L., Johansson, F., Zwahlen, M., von Heijne, G., Nielsen, J., and Ponten, F. (2015) Proteomics. Tissue-based map of the human proteome, Science 347, 1260419. Abstract/FREE Full Text Google Scholar
[7].↵Keen, J. C., and Moore, H. M. (2015) The Genotype-Tissue Expression (GTEx) Project: Linking Clinical Data with Molecular Analysis to Advance Personalized Medicine, J Pers Med
5, 22–29. Google Scholar
[8].↵Yu, N. Y., Hallstrom, B. M., Fagerberg, L., Ponten, F., Kawaji, H., Carninci, P., Forrest, A. R., Fantom, C., Hayashizaki, Y., Uhlen, M., and Daub, C. O. (2015) Complementing tissue characterization by integrating transcriptome profiling from the Human Protein Atlas and from the FANTOM5 consortium, Nucleic Acids Res 43, 6787–6798. CrossRef PubMed Google Scholar
[9].↵Uhlen, M., Karlsson, M. J., Zhong, W., Tebani, A., Pou, C., Mikes, J., Lakshmikanth, T., Forsstrom, B., Edfors, F., Odeberg, J., Mardinoglu, A., Zhang, C., von Feilitzen, K., Mulder, J., Sjostedt, E., Hober, A., Oksvold, P., Zwahlen, M., Ponten, F., Lindskog, C., Sivertsson, A., Fagerberg, L., and Brodin, P. (2019) A genome-wide transcriptomic analysis of protein-coding genes in human blood cells, Science 366. Google Scholar
[10].↵Uhlen, M., Hallstrom, B. M., Lindskog, C., Mardinoglu, A., Ponten, F., and Nielsen, J. (2016) Transcriptomics resources of human tissues and organs, Mol Syst Biol 12, 862.
Abstract/FREE Full Text Google Scholar
[11].↵Hamming, I., Timens, W., Bulthuis, M. L., Lely, A. T., Navis, G., and van Goor, H. (2004) Tissue distribution of ACE2 protein, the functional receptor for SARS coronavirus. A first step in understanding SARS pathogenesis, J Pathol 203, 631–637. CrossRef PubMed Web of Science Google Scholar
[12].↵Uhlen, M., Bandrowski, A., Carr, S., Edwards, A., Ellenberg, J., Lundberg, E., Rimm, D. L., Rodriguez, H., Hiltke, T., Snyder, M., and Yamamoto, T. (2016) A proposal for validation of antibodies, Nat Methods 13, 823–827. CrossRef PubMed Google Scholar
[13].↵Edfors, F., Hober, A., Linderback, K., Maddalo, G., Azimi, A., Sivertsson, A., Tegel, H., Hober, S., Szigyarto, C. A., Fagerberg, L., von Feilitzen, K., Oksvold, P., Lindskog, C., Forsstrom, B., and Uhlen, M. (2018) Enhanced validation of antibodies for research applications, Nat Commun 9, 4130. CrossRef PubMed Google Scholar
N., Goel, R., Khan, A. A., Ahmad, S., Dey, G., Mudgal, K., Chatterjee, A., Huang, T. C., Zhong, J., Wu, X., Shaw, P. G., Freed, D., Zahari, M. S., Mukherjee, K. K., Shankar, S., Mahadevan, A., Lam, H., Mitchell, C. J., Shankar, S. K., Satishchandra, P., Schroeder, J. T., Sirdeshmukh, R., Maitra, A., Leach, S. D., Drake, C. G., Halushka, M. K., Prasad, T. S., Hruban, R. H., Kerr, C. L., Bader, G. D., Iacobuzio-Donahue, C. A., Gowda, H., and Pandey, A. (2014) A draft map of the human proteome, Nature 509, 575–581. CrossRef PubMed Web of Science
Google Scholar
[15].↵Wilhelm, M., Schlegl, J., Hahne, H., Gholami, A. M., Lieberenz, M., Savitski, M. M., Ziegler, E., Butzmann, L., Gessulat, S., Marx, H., Mathieson, T., Lemeer, S., Schnatbaum, K., Reimer, U., Wenschuh, H., Mollenhauer, M., Slotta-Huspenina, J., Boese, J. H., Bantscheff, M., Gerstmair, A., Faerber, F., and Kuster, B. (2014) Mass-spectrometry-based draft of the human proteome, Nature 509, 582–587. CrossRef PubMed Web of Science Google Scholar
[16].↵Han, X., Zhou, Z., Fei, L., Sun, H., Wang, R., Chen, Y., Chen, H., Wang, J., Tang, H., Ge, W., Zhou, Y., Ye, F., Jiang, M., Wu, J., Xiao, Y., Jia, X., Zhang, T., Ma, X., Zhang, Q., Bai, X.,
Lai, S., Yu, C., Zhu, L., Lin, R., Gao, Y., Wang, M., Wu, Y., Zhang, J., Zhan, R., Zhu, S., Hu, H., Wang, C., Chen, M., Huang, H., Liang, T., Chen, J., Wang, W., Zhang, D., and Guo, G. (2020)
Construction of a human cell landscape at single-cell level, Nature . Google Scholar
[17].↵Reyfman, P. A., Walter, J. M., Joshi, N., Anekalla, K. R., McQuattie-Pimentel, A. C., Chiu, S., Fernandez, R., Akbarpour, M., Chen, C. I., Ren, Z., Verma, R., Abdala-Valencia, H., Nam, K., Chi, M., Han, S., Gonzalez-Gonzalez, F. J., Soberanes, S., Watanabe, S., Williams, K. J. N., Flozak, A. S., Nicholson, T. T., Morgan, V. K., Winter, D. R., Hinchcliff, M., Hrusch, C. L., Guzy, R. D., Bonham, C. A., Sperling, A. I., Bag, R., Hamanaka, R. B., Mutlu, G. M., Yeldandi, A. V., Marshall, S. A., Shilatifard, A., Amaral, L. A. N., Perlman, H., Sznajder, J. I., Argento, A. C., Gillespie, C. T., Dematte, J., Jain, M., Singer, B. D., Ridge, K. M., Lam, A. P., Bharat, A., Bhorade, S. M., Gottardi, C. J., Budinger, G. R. S., and Misharin, A. V. (2019) Single-Cell Transcriptomic Analysis of Human Lung Provides Insights into the Pathobiology of Pulmonary Fibrosis, Am J Respir Crit Care Med 199, 1517–1536. Google Scholar
[18].↵Viera Braga, F. K., G.; Berg, M.; Carpaij, OA.; Polanski, K.; Simon, LM.; Brouwer, S.; Gomes, T.; Hesse, L.; Jiang, J.; Fasouli, ES.; Efremova, M.; Vento-Tormo, R.; Talavera-López, C.; Jonker, MR.; Affleck, K.; Palit, S.; Strzelecka, PM.; Firth, HV.; Mahbubani, KT.; Cvejic, A.; Meyer, KB.; Saeb-Parsy, K.; Luinge, M.; Brandsma, C.; Timens, W.; Angelidis, I.; Strunz, M.; Koppelman, GH.; van Oosterhout, AJ.; Schiller, HB.; Theis, FJ.; van den Berge, M.; Nawijn, MC.; Teichmann, SA. (2019) A cellular census of human lungs identifies novel cell states in health and in asthma, Nat Med 25, 1153–1163. CrossRef Google Scholar
[19].↵Zou, X., Chen, K., Zou, J., Han, P., Hao, J., and Han, Z. (2020) Single-cell RNA-seq data analysis on the receptor ACE2 expression reveals the potential risk of different human organs vulnerable to 2019-nCoV infection, Front Med. Google Scholar
[20].↵Zhao, Y. Z., Z; Wang, Y.; Zhou, Y.; Ma, Y.; Zuo, W. (2020) Single-cell RNA expression profiling of ACE2, the putative receptor of Wuhan 2019-nCov, bioRxiv
https://doi.org/10.1101/2020.01.26.919985. Google Scholar
[21].↵Lukassen, S. C., R.L.; Trefzer, T.; Kahn, N.C.; Schneider, M.A.; Muley, T.; Winter, H.; Meister, M.; Veith, C.; Boots, A.W.; Hennig, B.P.; Kreuter, M.; Conrad, C.; Eils, R. (2020) SARS- CoV-2 receptor ACE2 and TMPRSS2 are predominantly expressed in a transient secretory cell type in subsegmental bronchial branches, bioRxiv https://doi.org/10.1101/2020.03.13.991455. Google Scholar
[23].↵Xu, H., Zhong, L., Deng, J., Peng, J., Dan, H., Zeng, X., Li, T., and Chen, Q. (2020) High expression of ACE2 receptor of 2019-nCoV on the epithelial cells of oral mucosa, Int J Oral Sci 12, 8. Google Scholar
[24].↵Zhang, H. K., Z.; Gong, H.; Xu, D; Wang, J.; Li, Z.; Cui, X.; Xiao, J.; Meng, T.; Zhou, W.; Liu, J.; Xu, H. (2020) The digestive system is a potential route of 2019-nCov infection: a bioinformatics analysis based on single-cell transcriptomes, bioRxiv https://doi.org/10.1101/2020.01.30.927806. Google Scholar
[25].↵Giard, D. J., Aaronson, S. A., Todaro, G. J., Arnstein, P., Kersey, J. H., Dosik, H., and Parks, W. P. (1973) In vitro cultivation of human tumors: establishment of cell lines derived from a series of solid tumors, J Natl Cancer Inst 51, 1417–1423. CrossRef PubMed Web of Science Google Scholar
[26].↵Thul, P. J., Akesson, L., Wiking, M., Mahdessian, D., Geladaki, A., Ait Blal, H., Alm, T., Asplund, A., Bjork, L., Breckels, L. M., Backstrom, A., Danielsson, F., Fagerberg, L., Fall, J., Gatto, L., Gnann, C., Hober, S., Hjelmare, M., Johansson, F., Lee, S., Lindskog, C., Mulder, J., Mulvey, C. M., Nilsson, P., Oksvold, P., Rockberg, J., Schutten, R., Schwenk, J. M., Sivertsson, A., Sjostedt, E., Skogs, M., Stadler, C., Sullivan, D. P., Tegel, H., Winsnes, C., Zhang, C., Zwahlen, M., Mardinoglu, A., Ponten, F., von Feilitzen, K., Lilley, K. S., Uhlen, M., and Lundberg, E. (2017) A subcellular map of the human proteome, Science 356. Google Scholar
[27].↵Xu, Y., Li, X., Zhu, B.; Liang, H.; Fang, C.; Gong, Y.; Guo, Q.; Sun, X.; Zhao, D.; Shen, J.; Zhang, H.; Liu, H.; Xia, H.; Tang, J.; Zhang, K.; Gong, S. (2020) Characteristics of pediatric SARS-CoV-2 infection and potential evidence for persistent fecal viral shedding., Nat Med. Google Scholar
[28].↵Kampf, C., Olsson, I., Ryberg, U., Sjostedt, E., and Ponten, F. (2012) Production of tissue microarrays, immunohistochemistry staining and digitalization within the human protein atlas, J Vis Exp. Google Scholar
